Eine Vision Zero in der Onkologie kann über einen Digitalisierungs-Masterplan die sichere Datennutzung und die Versorgung der Patient:innen entscheidend verbessern. Dies ist eine der zentralen Forderungen der „Berliner Erklärung“ des E-Health Summit 2021 von Vision Zero e.V.. Das größte Hindernis auf diesem Weg ist eigentlich nicht eine fehlende Bereitschaft zum Austausch von Daten, sondern die sehr unterschiedlichen sprachlichen Ausdrücke, Begriffe und Definitionen von Datenbankfeldern für inhaltlich ähnliche Informationen in den verschiedenen medizinischen Datenbeständen aus Forschung und Versorgung. Als Konsequenz hat Vision Zero begonnen, einen Prozess für die Definition einer gemeinsamen Datensprache anzustoßen, also einen umfassenden gemeinsamen Datenstandard aller gebräuchlichen Datenfelder, den German OncoLogical Data Standard (GOLD).
Das Datensatz-Team der Arbeitsgruppe konnte auf Erfahrungen aus dem Netzwerk Universitätsmedizin und der Medizininformatik-Initiative zurückgreifen. Hier wurden Prozesse etabliert, die in einem Kerndatensatz für die medizinische Forschung und in einem Konsensusdatensatz für die COVID-Forschung, dem sogenannten GECCO (German Corona Consensus Data Set), resultierten.
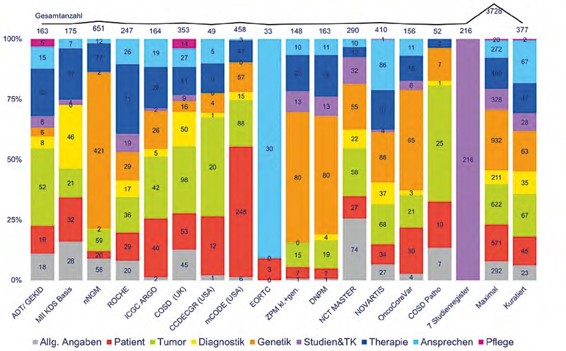
Inhaltliche Verteilung von Definitionen originaler (Meta-)Datenitems aus Formaten von 15 Datensätzen, 7 Studienregistern und der erstellten Maximaltabelle in 9 Domänen, angelehnt an eine onkologische Patient Journey
Der Datensatzkonsensus auf Basis einer onkologischen Patient Journey geschieht unter ähnlichen Gesichtspunkten, ist aber wesentlich komplexer. Der Umfang der zu berücksichtigenden Datenitems und Spezifikationen ist viel größer. In Vorbereitung auf GOLD wurde eine vergleichende Übersicht über die Datenitems wesentlicher gängiger Datensammlungen erstellt, und die Items wurden in sinnvolle Parametergruppen sortiert. Erste Harmonisierungen unterschiedlicher Daten- satzformate konnten bereits in Form von harmonisierten FHIR-Profilen für Datenelemente in Zusammenarbeit mit
z.B. der Medizininformatik-Initiative und DNPM (Netzwerk Personalisierte Medizin) erstellt werden.
Unsere Vision – was wollen wir erreichen?
Es wird kein neuer Datensatz entwickelt, sondern es werden bereits bestehende, fachlich abgestimmte Datensatzformate analysiert und harmonisiert. Daraus wird ein interoperabler Standard sowohl für die Versorgung als auch für die akademische und industrielle Forschung entwickelt. Der gesamte Patientenweg wird in konsentierten Datenformaten abgebildet.
Die Definitionen der Datenitems, also die in den Datensätzen verwendeten Datenformate, wurden in fachliche Gruppen geordnet und in einer gemeinsamen Sprache abgebildet. Auch die Datenstruktur und -formate sollten dabei standardisiert werden, sodass die Datenitems interoperabel sind. Auf diese Weise soll die Arbeit der Arbeitsgruppen Onkologie des Interop Councils wirksam unterstützt werden.
Durch die Harmonisierung und Verknüpfung der „zersplitterten“ Formate der Datensätze aus verschiedenen Schwerpunktbereichen sollen möglichst alle Definitionen von Datenitems über die komplette onkologische Patient Journey für den klinischen Alltag wie auch die akademische und industrielle Forschung integriert werden. Es werden dabei bewusst keine Vorgaben gemacht, ob und welche Daten in welchen Kontexten verpflichtend erhoben werden müssen, dies bleibt eine individuelle Entscheidung der Nutzenden. Ziel ist es, eine interoperable Definition zu jedem in der Praxis verwendeten Datenitem zu erstellen und auch strukturiert bereitzustellen. Das schafft optimale Voraussetzungen für die medizinische Behandlung aller Krebspatient:innen, Stichwort Präzisionsmedizin, für die (insbesondere translationale) Forschung sowie für Analysen mit Public-Health-Fokus.
Unsere Vorüberlegungen – was ist wichtig?
In Vorüberlegungen über die Herangehensweise zur Erstellung eines Konsensus bilden sich rasch die folgenden Ziele heraus:
1. Der inhaltliche Aufbau des Datenstandards muss die komplette onkologische Patient Journey logisch abbilden, zum Beispiel auch die in den meisten Datensätzen noch fehlenden molekularen Abfragen.
2. Eine Aktualisierung und Erweiterung muss entsprechend dem Stand der Forschung durchgehend möglich sein.
3. Der Krankheitsverlauf der Patient:innen muss longitudinal erfasst werden, also über den kompletten Behandlungsverlauf mit Verweis auf Zeit, Ort und Stadium.
4. Die Multiplizität, also das Abbilden von wiederkehrenden Ereignissen, muss zu allen Zeiten, auch bei einem Rezidiv nach mehreren Jahren, möglich sein.
5. Ein Versionierungsmanagement des Datenstandards und der integrierten Kataloge wie SNOMED oder ICD-10 Codesysteme ist zwingend nötig.
6. Der Datenstandard muss auf bereits vorhandenen interoperablen Spezifikationen, insbesondere HL7® FHIR®, aus Definitionen anderer Daten(an)sätze aufbauen.
Unser Vorgehen – was haben wir bisher gemacht?
Inhaltliche Aufbereitung – Aufbau auf Vorhandenem
Als Ausgangspunkt für dieses Projekt wurden 15 wichtige in Deutschland und international verwendete Datensatzformate ausgewählt und deren Originalitems ohne Informationsverlust zu einer sogenannten Maximaltabelle zusammengefügt, in der alle Datenitem-Definitionen enthalten sind.
Die bisher eingeflossenen Formate von 15 Datensätzen und 7 Studienregistern umfassen neben deutschen Datendefinitionen, wie den gesetzlich verankerten onkologischen Basisdatensatz ADT/GEKID, auch weitere aus den USA, Großbritannien und dem europäischen Raum. Bei der Zusammenführung der Definitionen fiel schnell auf, dass selbst bei einfachsten Inhalten keine sprachliche Einheitlichkeit vorliegt und dass keines der Datensatzformate eine gesamte Patient Journey abbilden kann.
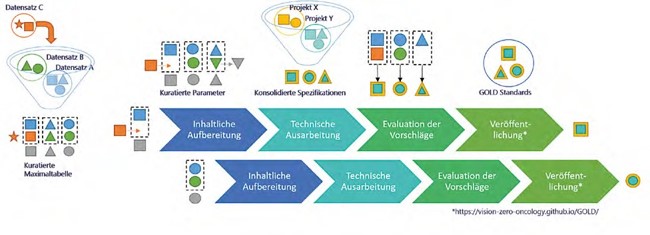
Darstellung des GOLD-Prozesses und der iterativen VorgehensweiseDarstellung des GOLD-Prozesses und der iterativen Vorgehensweise
Um eine Vergleichbarkeit zwischen den Datensatzformaten erreichen zu können, wurden die mehr als 3700 originalen Datenitem-Definitionen in der Maximaltabelle zu aktuell 9 Domänen und 377 vorkuratierten Datenparametergruppen mit im Durchschnitt etwa 10 Datenformatitems manuell zugeordnet, um inhaltlich gleiche Aspekte zusammenzufassen. Die Abbildung 1 zeigt die inhaltliche Verteilung der Domänen in den verschiedenen Quelldatensatzformaten. Die unterschiedlichen Fokussierungen sorgen für thematisch vielfältige und ausgeglichene Datenitem-Definitionen in der Maximaltabelle. Die Auswahl der Quelldatensatzformate ist nicht als endgültig zu betrachten. Auch die Aufwandsabschätzung zeigt, dass noch etliche Datenitems für die komplette Patient Journey fehlen. Momentan wird an einer Automatisierung der Prozesse zur inhaltlichen Aufbereitung gearbeitet, um in Zukunft sowohl neue Datensatzformate hinzufügen als auch die bereits enthaltenen Datensatzformate up to date halten zu können.
Mit der beschriebenen Kuratierung wurde die Grundlage für den weiteren Prozess in Richtung der Erstellung eines gemeinsamen Datenstandards gelegt. Hierbei ist das Nutzungspotenzial der kuratierten Parameter nicht nur auf die Onkologie beschränkt. Zwei Drittel der Parameter können auch über die Onkologie hinaus verwendet werden, wodurch diese Arbeit generell für die Versorgung einen deutlichen Mehrwert darstellen wird.
Technische Ausarbeitung – als nächster Schritt FHIR
Im weiteren Verlauf wurde, basierend auf den inhaltlichen Ergebnissen, die technische Ausarbeitung dieser Information begonnen. Dabei wurden erste kuratierte Parametergruppen mit den enthaltenen originalen Datenitem-Definitionen ausgewählt, um nach vorhandenen FHIR-Spezifikationen aus anderen Projekten zu suchen, die diese Inhalte abbilden. Eine Auswahl von Projekten, deren FHIR-Profile als Grundlage verwendet wurden, sind:
• ISiK (Informationstechnische Systeme in Krankenhäusern) als verbindlicher Standard für den Austausch von der gematik GmbH
• Onkologische Basisprofile von HL7 Deutschland e.V.
• MII KDS (Kerndatensatz der Medizininformatik-Initiative)
• nNGM (Datensatz des nationalen Netzwerks Genomische Medizin)
• mCode (Minimal Common Oncology Data Elements)
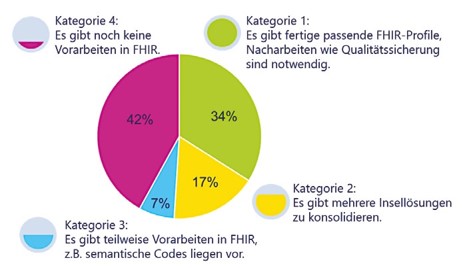
Erfahrungswerte zur Vollständigkeit der FHIR- Spezifikationen, die basierend auf den Datenitem- Definitionen der Maximaltabelle erstellt werden sollen
Die Eigenschaften aller gefundenen FHIR-Spezifikationen wurden einander gegenübergestellt und verglichen. Oftmals gab es für die gleiche Parametergruppe mehrere FHIR- Profile, die nicht immer miteinander konform waren. So ist beispielsweise die TNM-Klassifikation UICC in mindestens 8 Quelldatensätzen mit 6 FHIR-Spezifikationen erfasst. Bei dem Vergleich der bestehenden FHIR-Profile wurde auf standardisierte semantische und in Deutschland verwendete Codes wie LOINC und SNOMED CT geachtet. Aufbauend auf den vorhandenen Profilen, insbesondere bei verbindlichen Standards (ISiK), wurde ein GOLD-Vorschlag erarbeitet, der inhaltlich vollständig und möglichst kompatibel mit anderen Projekten ist. Als Beispiel wurde für das histopathologische Grading der WHO durch das GOLD-Projekt eine Erweiterung um die SNOMED-Codes vorgeschlagen.
Die inhaltliche Aufbereitung und technische Ausarbeitung erfolgt durch die Umsetzungsgruppe, die sich aus dem BIH / Charité-Team, Forscher:innen und praktizierenden Onkolog:innen des NCT Heidelberg und des CCC München (LMU), Interoperabilitätsspezialist:innen und Softwareentwicker:innen der mint medical GmbH sowie Vertreter:innen der Roche Pharma AG und Novartis Pharma GmbH zusammensetzt.
Evaluation – Verabschiedung der GOLD-FHIR-Vorschläge
Um einem gemeinsamen Konsens näher zu kommen, wurde zu Projektbeginn eine Evaluierungsgruppe gegründet, welche relevante Akteure aus unterschiedlichen Bereichen des Gesundheitssystems zusammenbrachte. Die Evaluierungsgruppe bestand sowohl aus Mitgliedern der oben genannten Umsetzungsgruppe als auch aus weiteren Stakeholdern aus den Bereichen der medizinischen und klinischen Forschung und Medizininformatik. In dieser Runde wurden die ersten Arbeitsergebnisse der Umsetzungsgruppe inklusive der erarbeiteten Harmonisierungsvorschläge vorgestellt und über ein eigens erstelltes Umfragetool in REDCap zur Abstimmung freigegeben. Durch die Einberufung des bei der gematik angesiedelten Interop Councils in Folge eines noch auf Jens Spahn zurückgehenden Bundesgesetzes im Dezember 2021 gibt es inzwischen ein Gremium, zu dessen Aufgaben es gehört, die Verwendung von verbindlichen Standards im Gesundheitswesen voranzubringen. Somit können die von GOLD erarbeiteten Vorschläge für Standards zur Evaluation direkt ins Interop Council eingebracht werden.
Veröffentlichungen – nur veröffentlichte Vorschläge können genutzt werden
Mit dem beschriebenen Prozess (s. Abbildung 2) ist es gelungen, für fast 20 Prozent der Maximaltabelle GOLD-FHIR- Vorschläge zu entwickeln. Diese wurden im engen Austausch von HL7 Deutschland finalisiert und als Onkologische Basisprofile qualitätsgesichert im Interoperabilitäts- Navigator (INA) der gematik veröffentlicht. Die bisher veröffentlichten FHIR-Vorschläge sind unter der Adresse https://vision-zero-oncology.github.io/GOLD/ einsehbar.
Ausblick – was wir noch brauchen
Unser Ziel ist nicht die weitere Definition einer Teilmenge der krebsbezogenen Daten, z. B. für einen Use Case, denn solche Insellösungen gibt es bereits mehr als genug. Die Digitalisierung der Medizin wird nicht allein mit Provisorien und innerhalb von Projekten gelingen. Stattdessen sollten wir das Gesamtbild mit aller Komplexität im Blick behalten und streben daher eine natürlich schrittweise Standardisierung aller Datenfelder der gesamten Real-World Patient Journey auf nationaler Ebene für Versorgung, Forschung und Public Health (Register) an. Die gesammelten Erfahrungen mit dem End-to-End Konsens-Prozess von den ersten Datenitem- Definitionen zu den Vorschlägen für FHIR-Profile erlauben uns interessanterweise eine datenbasierte Aufwandsschätzung. Darin ist der zeitliche und finanzielle Aufwand dargestellt, um eine komplette onkologische Patient Journey (extrapoliert auf etwa 500 kuratierte Parametergruppen) in FHIR umzusetzen. Es wurde auch der Erfahrungswert zur Vollständigkeit der bestehenden FHIR-Spezifikationen miteinbezogen. Für etwa 34 Prozent der kuratierten Parametergruppen könnten zum Zeitpunkt der Schätzung vorliegende Spezifikationen als deutschlandweite Standards übernommen werden, während bei fast der Hälfte der Datenelemente größere Anpassungen vorhandener Spezifikationen empfohlen werden bzw. eine Neuprofilierung notwendig ist (s. Abbildung 3).
Auch wenn die Aufwandsschätzung eine ungefähre Anzahl an kuratierten Parametergruppen kalkuliert, ist das GOLD-Projekt selbst nach der Standardisierung aller Parameter nicht beendet. Außerdem müssen diese dann von den Softwareherstellern getestet und implementiert werden. Zudem ist es wichtig, schon von Beginn an den Standardisierungsprozess von neuen Datenitems mitzudenken. Die Medizin entwickelt sich rasant weiter. Auch durch die großen Fortschritte in der Datenanalyse, insbesondere im molekularen Bereich, ist davon auszugehen, dass in Zukunft innerhalb kürzester Zeit zahlreiche neue Datenitems entwickelt werden, für die Standards festgelegt werden müssen, z. B. neue treibende Genmutationen, neue Tumormarker, neue diagnostischen Methoden, neue Immuntherapeutika. Um diesen Ansprüchen gerecht zu werden, muss für alle verwendeten Definitionen von Datenitems eine agile Weiterentwicklung der Standards möglich sein, in der auch die vorläufige Definition von neuen Datenitems sofort eingebracht und weitergetragen werden kann. Dabei müssen die Inhalte von medizinischen Expert:innen und Forschenden effizient definiert und abgestimmt werden können. Diese Funktionalitäten müssen von einer zentralen Meldestelle, am besten auch vorgefertigt in den Formaten der häufigsten Anwendungen, bereitgestellt werden. Dies sollte national durch den Interoperabilitäts-Navigator INA der gematik gewährleistet und politisch durchgesetzt werden. Das heißt, dass Softwareanbieter diese Spezifikationen zwingend umzusetzen haben.
Ein gemeinsames Ziel: Interop Council und GOLD-Projekt Die Notwendigkeit eines einheitlichen Standards beziehungsweise einer gemeinsamen Datensprache war einer der Gründe zur Initiierung des GOLD-Projekts im Spätsommer 2021. Im Dezember 2021 hat die Koordinierungsstelle Interoperabilität der gematik in Abstimmung mit dem Bundesgesundheitsministerium als Mehrheitsgesellschafter ein nationales Expertengremium für Interoperabilität, das Interop Council, einberufen. Die Leitung dieses Gremiums liegt bei Prof. Dr. Sylvia Thun, die gemeinsam mit ihrem Team seit Beginn am GOLD-Projekt beteiligt ist. Das Gremium hat bereits erste Empfehlungen, beispielsweise zur deutschlandweiten Nutzung von FHIR R4 im Sommer 2022, ausgesprochen. Die Diskussionen machen deutlich, dass FHIR alleine kein Wundermittel ist. Es ist zwar für Interoperabilität unabdingbare Voraussetzung, aber viele vor allem prozessuale und organisatorische Fragen bleiben offen.
Um dem gemeinsamen Ziel, mit einem interoperablen Datenstandard alle Akteure im Gesundheitssystem deutlich voranzubringen, stellen wir sowohl die Expertise des GOLD- Projekts, also involvierte Expert:innen, als auch die bisherigen Vorarbeiten auf inhaltlicher und technischer Ebene (inkl. Aufwandsschätzung) für die Arbeit des Interop Council zur Verfügung.
Wir danken allen Unterstützer:innen für ihre tatkräftige Mitarbeit. Unser besonderer Dank gilt dabei den Mitgliedern der GOLD Umsetzungs- und Evaluierungsgruppe. Kontakt: gold@charite.de
Wir brauchen eine gemeinsame Datensprache – und zwar jetzt! Spätestens seit der COVID-19-Pandemie ist klar geworden, dass Daten tatsächlich Leben retten können. Das GOLD-Projekt (German OncoLogical Data Standard) will durch eine möglichst vollständige Standardisierung der Gesundheitsdaten die Behandlung von Krebspatient:innen verbessern. Dabei setzt GOLD auf die Verwendung und ggf. Weiterentwicklung von bereits bestehenden Inhalten wie Definitionen von Datenitems und FHIR- Spezifikationen, die mit viel Aufwand zusammengetragen, analysiert und gegebenenfalls ergänzt wurden, um daraus den Vorschlag für einen interoperablen Standard zu entwickeln. Das GOLD-Projekt wurde in den letzten beiden Jahren mit viel Engagement aller Beteiligten, jedoch ohne Finanzierung vorangetrieben. Wir sind an eine Skalierbarkeitsgrenze gestoßen. Wir arbeiten an einer Projektfinanzierung durch die beteiligten Partner, möglicherweise als Public-private-Partnership, um innerhalb von zwei Jahren HL7® FHIR®-Vorschläge für die komplette onkologische Patient Journey zu entwickeln. Wir haben die Chance, einen tatsächlichen Mehrwert zu schaffen, und das deutlich über Onkologie hinaus. Durch das Interop Council gibt es jetzt die Möglichkeit, die Vorschläge für FHIR-Spezifikationen zu evaluieren und tatsächliche Standards zu erstellen, die Erkenntnisse durch ein lernendes Gesundheitssystem und damit auch die Versorgung der Patient:innen auf ein neues Level heben werden.
































